Drosha
Drosha是一種RNA酶III[5],在人類基因組中由5號染色體上的DROSHA基因(舊稱RNASEN)編碼[6][7][8],於2000年被克隆發表,最初被發現為切割rRNA前驅物(pre-rRNA)的一種RNA酶[9],現已知其主要功能為在miRNA生成的初期切割miRNA的前驅物,此蛋白可與DGCR8蛋白組成微加工复合体[10],將DNA轉錄產生的pri-miRNA切割成長約70nt的pre-miRNA,後者可再由Dicer切割產生成熟的miRNA[11]。Drosha、Dicer與其他參與miRNA生成的蛋白之表現量與某些癌症相關[12]。
功能
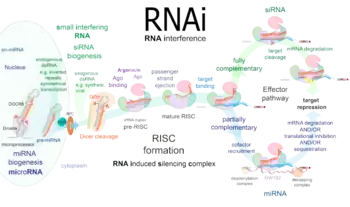

RNA酶III皆為切割雙股RNA的RNA內切酶,其中Drosha在細胞核中參與miRNA前驅物切割的初始步驟[8][11]。miRNA的生成過程最初是由RNA聚合酶II轉錄產生可長達數kb、具5′端帽與多腺苷酸尾的初級轉錄本pri-miRNA(初級miRNA)[13][14],其受Drosha切割後會形成長約70nt、且3′端具2個突出鹼基(overhang)的pre-miRNA(前miRNA)。pre-miRNA可與XPO5蛋白結合,由細胞核被送入細胞質中,其3′端的突出鹼基可被另一種RNA酶IIIDicer所識別,後者可再將pre-miRNA切割成長22nt的雙股RNA,其中的一股即是成熟的miRNA,可與RNA誘導沉默複合體(RISC)結合而進行RNA干擾,切割目標mRNA或抑制其轉譯以達成基因靜默的效果[15]。
Drosha切割pri-miRNA時會與兩個RNA結合蛋白DGCR8共同組成稱為微加工复合体的蛋白三聚體[16][17][18][19],DGCR8在模式生物黑腹果蠅與秀麗隱桿線蟲中稱為Pasha,即「Drosha的夥伴蛋白」(partner of Drosha)之簡稱[20],Drosha需在與DGCR8結合的情況下才能進行切割[21]。除必要的Drosha與DGCR8外,微加工复合体還可能包含EWSR1、异质核糖核蛋白、FUS與DEAD-BoxRNA解旋酶(p68、p72)等其他蛋白以幫助切割pri-miRNA[22][23],有些種類的pri-miRNA只有在特定輔助蛋白存在時才能被Drosha切割[24]。
Drosha大多位於細胞核中,但也有些Drosha不含核定位序列(NLS)而位於細胞質中,稱為c-Drosha,可能以其他機制調控基因表現[25][26]。另外Drosha與Dicer也參與DNA修補[27]。
少數miRNA以型的方式生成,不需經Drosha切割,此類miRNA稱為Mirtron,編碼序列位於其他基因的內含子中,可隨該基因的mRNA轉錄後進行剪接時被切割形成pre-miRNA,因此不需依賴Drosha[28];此外,還有些miRNA(simtron)前驅物的切割仰賴Drosha,但不需DGCR8、XPO5與Dicer[29]。
臨床意義
Drosha等參與miRNA生成的蛋白表現量與某些癌症相關[12],例如某些種類的乳癌病患的Drosha與Dicer的表現量下降[30],癌症基因組圖譜中也顯示數種乳癌、大腸癌與食道癌病患細胞質中的Drosha(即c-Drosha)表現量增加[25]。
參考文獻
- GRCh38: Ensembl release 89: ENSG00000113360 - Ensembl, May 2017
- GRCm38: Ensembl release 89: ENSMUSG00000022191 - Ensembl, May 2017
- . National Center for Biotechnology Information, U.S. National Library of Medicine.
- . National Center for Biotechnology Information, U.S. National Library of Medicine.
- Filippov V, Solovyev V, Filippova M, Gill SS. . Gene. 2000, 245 (1): 213–21. PMID 10713462. doi:10.1016/s0378-1119(99)00571-5.
- Filippov V, Solovyev V, Filippova M, Gill SS. . Gene. 2000, 245 (1): 213–21. PMID 10713462. doi:10.1016/S0378-1119(99)00571-5.
- Wu H, Xu H, Miraglia LJ, Crooke ST. . The Journal of Biological Chemistry. 2000, 275 (47): 36957–65. PMID 10948199. doi:10.1074/jbc.M005494200
 .
. - .
- Wu H, Xu H, Miraglia LJ, Crooke ST. . J Biol Chem. 2000, 275 (47): 36957–65. PMID 10948199. doi:10.1074/jbc.M005494200.
- Denli AM, Tops BB, Plasterk RH, Ketting RF, Hannon GJ. . Nature. 2004, 432 (7014): 231–5. PMID 15531879. S2CID 4425505. doi:10.1038/nature03049.
- Lee Y, Ahn C, Han J, Choi H, Kim J, Yim J, Lee J, Provost P, Rådmark O, Kim S, Kim VN. . Nature. 2003, 425 (6956): 415–9. PMID 14508493. S2CID 4421030. doi:10.1038/nature01957.
- Slack FJ, Weidhaas JB. . N Engl J Med. 2008, 359 (25): 2720–2. PMID 19092157. doi:10.1056/NEJMe0808667.
- Conrad T, Ntini E, Lang B, Cozzuto L, Andersen JB, Marquardt JU; et al. . RNA. 2020, 26 (11): 1726–1730. PMC 7566579
 . PMID 32669295. doi:10.1261/rna.076240.120.
. PMID 32669295. doi:10.1261/rna.076240.120. - Cai X, Hagedorn CH, Cullen BR. . RNA. 2004, 10 (12): 1957–66. PMC 1370684
 . PMID 15525708. doi:10.1261/rna.7135204.
. PMID 15525708. doi:10.1261/rna.7135204. - Saito K, Ishizuka A, Siomi H, Siomi MC. . PLoS Biol. 2005, 3 (7): e235. PMC 1141268
 . PMID 15918769. doi:10.1371/journal.pbio.0030235.
. PMID 15918769. doi:10.1371/journal.pbio.0030235. - Partin, Alexander C.; Zhang, Kaiming; Jeong, Byung-Cheon; Herrell, Emily; Li, Shanshan; Chiu, Wah; Nam, Yunsun. . Molecular Cell. 2020, 78 (3): 411–422.e4. PMC 7214211
 . PMID 32220646. doi:10.1016/j.molcel.2020.02.016.
. PMID 32220646. doi:10.1016/j.molcel.2020.02.016. - Kwon SC, Nguyen TA, Choi YG, Jo MH, Hohng S, Kim VN, Woo JS. . Cell. 2016, 164 (1–2): 81–90. PMID 26748718. doi:10.1016/j.cell.2015.12.019
 .
. - Herbert KM, Sarkar SK, Mills M, Delgado De la Herran HC, Neuman KC, Steitz JA. . RNA. 26683315, 22 (2): 175–83. PMC 4712668
 . doi:10.1261/rna.054684.115.
. doi:10.1261/rna.054684.115. - Nguyen TA, Jo MH, Choi YG, Park J, Kwon SC, Hohng S, et al. . Cell. 2015, 161 (6): 1374–87. PMID 26027739. doi:10.1016/j.cell.2015.05.010
 .
. - Denli AM, Tops BB, Plasterk RH, Ketting RF, Hannon GJ. . Nature. 2004, 432 (7014): 231–5. PMID 15531879. doi:10.1038/nature03049.
- Han J, Lee Y, Yeom KH, Nam JW, Heo I, Rhee JK, Sohn SY, Cho Y, Zhang BT, Kim VN. . Cell. 2006, 125 (5): 887–901. PMID 16751099. S2CID 453021. doi:10.1016/j.cell.2006.03.043
 .
. - Siomi H, Siomi MC. . Molecular Cell. 2010, 38 (3): 323–32. PMID 20471939. doi:10.1016/j.molcel.2010.03.013
 .
. - Suzuki HI, Miyazono K. . J Biochem. 2011, 149 (1): 15–25. PMID 20876186. doi:10.1093/jb/mvq113.
- Ha M, Kim VN. . Nature Reviews. Molecular Cell Biology. 2014, 15 (8): 509–24. PMID 25027649. S2CID 205495632. doi:10.1038/nrm3838.
- Dai L, Chen K, Youngren B, Kulina J, Yang A, Guo Z; et al. . Nucleic Acids Res. 2016, 44 (21): 10454–10466. PMC 5137420
 . PMID 27471035. doi:10.1093/nar/gkw668.
. PMID 27471035. doi:10.1093/nar/gkw668. - Link S, Grund SE, Diederichs S. . Nucleic Acids Research. 2016, 44 (11): 5330–43. PMC 4914122
 . PMID 27185895. doi:10.1093/nar/gk400.
. PMID 27185895. doi:10.1093/nar/gk400. - Francia S, Michelini F, Saxena A, Tang D, de Hoon M, Anelli V, Mione M, Carninci P, d'Adda di Fagagna F. . Nature. 2012, 488 (7410): 231–5. PMC 3442236
 . PMID 22722852. doi:10.1038/nature11179.
. PMID 22722852. doi:10.1038/nature11179. - Ruby, JG; Jan, CH; Bartel, DP. . Nature. 2007, 448 (7149): 83–6. Bibcode:2007Natur.448...83R. PMC 2475599
 . PMID 17589500. doi:10.1038/nature05983.
. PMID 17589500. doi:10.1038/nature05983. - Havens MA, Reich AA, Duelli DM, Hastings ML. . Nucleic Acids Res. 2012, 40 (10): 4626–40. PMC 3378869
 . PMID 22270084. doi:10.1093/nar/gks026.
. PMID 22270084. doi:10.1093/nar/gks026. - Thomson JM, Newman M, Parker JS, Morin-Kensicki EM, Wright T, Hammond SM. . Genes & Development. 2006, 20 (16): 2202–7. PMC 1553203
 . PMID 16882971. doi:10.1101/gad.1444406.
. PMID 16882971. doi:10.1101/gad.1444406.