奇異酵母
奇異酵母[2](學名:Saccharomyces paradoxus)是子囊菌門酵母屬的一種真菌,最早於1914年自俄羅斯的落葉樹樣本中發現,一度被視為釀酒酵母的變型或異名,後因分子證據而被確定為酵母屬中的獨立物種。本種酵母菌可分為數個演化支,分別分布於歐洲、北美洲、東亞等地,且彼此間已產生一定程度的生殖隔離,可能正在進行種化。
| 奇異酵母 | |
|---|---|
| 科学分类 | |
| 界: | 真菌界 Fungi |
| 门: | 子囊菌门 Ascomycota |
| 纲: | 酵母菌綱 Saccharomycetes |
| 目: | 酵母菌目 Saccharomycetales |
| 科: | 酵母菌科 Saccharomycetaceae |
| 属: | 酵母屬 Saccharomyces |
| 种: | 奇異酵母 S. paradoxus |
| 二名法 | |
| Saccharomyces paradoxus Bach.-Raich., 1914 | |
| 異名[1] | |
| |
奇異酵母為未經人類馴化與人擇選汰的野生酵母菌,一般僅見於溫帶地區野外落葉樹的樣本中,不見於人為發酵的酒或食物中;本種為與模式生物釀酒酵母關係最近的物種[3],兩者在外形與生理上都非常相似,惟本種的最適生長溫度稍低於釀酒酵母。
分類
奇異酵母最早於1914年由任職於彼得格勒女子醫學院(今聖彼得堡國立巴甫洛夫醫科大學)的俄羅斯真菌學家A. A. 巴钦斯卡娅-拉伊琴科( / A. A. Bachinskaya-Raichenko)發表描述,自聖彼得堡植物園的夏櫟以及波爾塔瓦州的歐洲野榆樣本中發現[4][5]。同年马丁努斯·威廉·拜耶林克也在荷蘭的橡樹樣本發現相似的酵母菌,將其發表為接合酵母屬的新種Zygosaccharomyces tetraspora。後續學者多將這兩個物種視為釀酒酵母的變型或異名。1989年,這兩個物種基於分子證據被視為同種,並自釀酒酵母中分出,重新被發表為酵母屬的獨立物種奇異酵母(Saccharomyces paradoxus)[6]。
奇異酵母的種小名paradoxus得名自發表者巴钦斯卡娅-拉伊琴科等早期學者認為本種的生活史有特別之處,即其子囊中的四個子囊孢子可共同融合而形成一個結構複雜的合子。但後來的學者並沒有發現此現象,奇異酵母的生活史與釀酒酵母並無相異之處,合子一樣是由兩個子囊孢子融合而成,且不同交配型的孢子才可融合產生合子[7]。
生理
奇異酵母是一種野生酵母菌,幾乎沒有自人為發酵的酒或食物中發現的紀錄[8]。本種一般見於橡樹、楓樹與樺樹等落葉樹的樣本中,也有自果蠅等昆蟲樣本中發現的紀錄[9][10][11][12],有時與釀酒酵母等同屬其他種酵母菌一起出現[13][14][15]。
奇異酵母與釀酒酵母的外形、生活史、以及發酵能力等生理特徵幾乎沒有差異,全基因組序列相似度約為88%[5],兩者約於距今500萬至1000萬年前分家[16]。兩物種在野外的共存模式與生態棲位差異仍有待更多研究闡明[17]。野生的奇異酵母與釀酒酵母菌株多無發酵麥芽糖的能力,奇異酵母的發表者巴钦斯卡娅-拉伊琴科即指出奇異酵母無法發酵麥芽糖。在人為培養之下,兩者皆可逐漸發生變異與適應,漸獲得發酵麥芽糖的能力[7]。
奇異酵母與釀酒酵母一樣為中溫生物,而酵母屬其他物種都較適合在低溫下生長,因此耐熱能力應為這兩個物種的共衍徵[18]。但奇異酵母的最適生長溫度又比釀酒酵母稍低,分布範圍也包括比釀酒酵母分布範圍冷的地區[9][15][3]。奇異酵母的最適生長溫度為攝氏37至38度,相較之下釀酒酵母則為41至42度,有研究表明北美洲的高緯度地區野外少有釀酒酵母分布,奇異酵母則相當常見;也有研究表明歐洲樣本中出現奇異酵母的機率和氣溫呈負相關;中國北方與南方均有釀酒酵母分布,奇異酵母則幾乎不見於南方的副熱帶和熱帶氣候區[19]。
奇異酵母大都行無性生殖,在環境中大多為二倍體(2N),有性生殖也有很高的比例是發生在同一子囊的孢子之間[20]。奇異酵母的菌株大多為同型合子,僅有WX21為異型合子(應是由兩菌株雜交產生)[19]。奇異酵母與釀酒酵母間有生殖隔離,雜交後僅有1%至3%可產生可萌發的孢子[21],且這些子代孢子通常帶有染色體數目的異常[5]。但仍有奇異酵母與釀酒酵母雜交產生的菌株在野外被發現[22],另外也有發現奇異酵母和另一同屬物種库德里阿兹威氏酵母雜交產生的菌株[23]。
族群演化
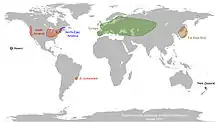
奇異酵母因未受人擇選汰影響,其生物地理分布反映了地理隔離與對環境因子的適應[10][25]。奇異酵母最初可細分為四大主要演化支,分別為分布於歐洲(包括西西伯利亞)和北美洲的SpA、分布於遠東(日本與東西伯利亞)的一個演化支、分布於北美洲(東西海岸與五大湖區)的SpB以及分布於北美東北部(加斯佩半島、聖羅倫斯河谷地與阿帕拉契山脈北緣)的SpC[3][10][25][26][27],四個株系彼此間已發生一定程度的合子後生殖隔離[28][29],可能正在經歷種化的過程[23]。此外夏威夷有一樣本可能構成第五個奇異酵母的演化支[10][26]。有些歐洲的菌株(SpA)在北美與紐西蘭出現,但在當地的序列多樣性都很低,可能是近代隨著外來種橡樹由殖民者引入[30][31]。隨著北美洲更多奇異酵母菌株的發現,北美族群共可細分為SpA、SpB、SpC、SpC*、SpD1和SpD2等6個子支序,其中SpC*可能是由SpB和SpC雜交而成的菌株衍生,SpD1和SpD2則可能是由SpC*和SpB雜交而成的菌株衍生[19][27],SpD1、D2已經和親本的SpB和SpC發生生殖隔離,SpC*則尚無[27]。中國的奇異酵母菌株多屬遠東支序,但有少數幾個菌株和遠東支序關係較遠而自成一群,且兩者的分布範圍沒有差異。因歐亞大陸的奇異酵母菌株多樣性遠高於北美,奇異酵母應是起源自歐亞大陸,且可能是起源自有兩個支序同時並存的中國[19]。
另外,南美洲有兩個原被認為是奇異酵母的樣本,因分子證據顯示其與本種序列有一定差異,已被描述為新種里約酵母[32],但有後續研究認為兩者關係緊密,尚未發生種化[29]。里約酵母與奇異酵母的序列差異不大,應屬奇異酵母北美支序中的SpB演化支[19],但兩者雜交後有生殖隔離,可萌發孢子的機率很低,這是因里約酵母有四段染色體發生易位所致[5]。
有研究顯示歐洲的奇異酵母支系有發生來自釀酒酵母的基因滲入[29];相對地也有奇異酵母的序列片段經基因滲入進入釀酒酵母菌株的基因組[33]。
應用
釀酒酵母被人類馴化、受人擇選汰已久,奇異酵母則未受此影響,可能更能反映野生酵母菌的生態,因此在一些演化與群體基因組學研究中被用作研究對象[26][34][35]。奇異酵母的基因組已於2003年被完整定序[36]。
奇異酵母雖與釀酒酵母關係接近,但奇異酵母為一種野生酵母,幾乎只見於自野外樹皮、落葉、土壤與昆蟲身上採集的樣本[5],鮮少在食品工業中被使用,也幾無在人為發酵的酒或食物中發現的紀錄[8],造成此現象的原因可能是奇異酵母較不適於生長在高溫的環境[19]。有研究指奇異酵母可能有被用於釀酒的潛力,且可能為酒帶來不同的風味[37][38][39],目前已有奇異酵母與釀酒酵母的雜交株被用於釀酒[40][41]。
參考文獻
- Cletus Kurtzman, J.W. Fell (编). . Elsevier Science. 1998: 366. ISBN 9780080542690.
- 龙思宇、严少敏. . 科学技术与工程. 2014, 14 (2).
- Johnson, LJ; Koufopanou, V; Goddard, MR. . Genetics. 2004, 166 (1): 43–52. PMC 1470673
 . PMID 15020405. doi:10.1534/genetics.166.1.43.
. PMID 15020405. doi:10.1534/genetics.166.1.43. - Batschinskaya, A.A. [History of development and culture of a new yeast fungus, Saccharomyces paradoxus]. J. Microbiol. Epidemiol. Immunobiol. 1914, 1: 231–247 (德语).
- Ono, Jasmine; Greig, Duncan; Boynton, Primrose J. . Annual Review of Microbiology. 2020-09-08, 74 (1): 477–495. doi:10.1146/annurev-micro-021320-014036.
- Martini, Ann Vaughan. . Systematic and Applied Microbiology. 1989-10, 12 (2): 179–182. doi:10.1016/S0723-2020(89)80012-8.
- Naumov, G. I. . Microbiology. 2013-07, 82 (4): 397–403. doi:10.1134/S0026261713040073.
- Louis, Edward J. . Fungal Biology Reviews. 2011-10, 25 (3): 136–142. doi:10.1016/j.fbr.2011.06.001.
- Charron, G; Leducq, J-B; Bertin, C. . FEMS Yeast Research. 2014, 14 (2): 281–8. PMID 24119009. doi:10.1111/1567-1364.12100
 .
. - Hyma, KE; Jay, JC. . Molecular Ecology. 2013, 22 (11): 2917–30. PMC 3620907
 . PMID 23286354. doi:10.1111/mec.12155.
. PMID 23286354. doi:10.1111/mec.12155. - Maganti, H; Bartfai, D; Xu, J. . FEMS Yeast Research. 2012, 12 (1): 9–19. PMID 22029478. doi:10.1111/j.1567-1364.2011.00756.x
 .
. - Sniegowski, PD; Dombrowski, PG; Fingerman, E. . FEMS Yeast Research. 2002, 1 (4): 299–306. PMID 12702333. doi:10.1111/j.1567-1364.2002.tb00048.x
 .
. - Naumov, GI; Naumova, ES; Sniegowski, PD. . Canadian Journal of Microbiology. 1998, 44 (11): 1045–50. PMID 10029999. doi:10.1139/w98-104.
- Sampaio, JP; Goncalves, P. (PDF). Applied and Environmental Microbiology. 2008, 74 (7): 2144–52. Bibcode:2008ApEnM..74.2144S. PMC 2292605
 . PMID 18281431. doi:10.1128/AEM.02396-07.
. PMID 18281431. doi:10.1128/AEM.02396-07. - Sweeney, JY; Kuehne, HA; Sniegowski, PD. . FEMS Yeast Research. 2004, 4 (4–5): 521–5. PMID 14734033. doi:10.1016/s1567-1356(03)00171-5
 .
. - Dori-Bachash, Mally; Shema, Efrat; Tirosh, Itay. . PLoS Biology. 2011-07-19, 9 (7): e1001106. doi:10.1371/journal.pbio.1001106.
- Sniegowski, Paul D; Dombrowski, Peter G; Fingerman, Ethan. . FEMS Yeast Research. 2002-01, 1 (4): 299–306. doi:10.1111/j.1567-1364.2002.tb00048.x.
- Peris, David; Ubbelohde, Emily J.; Kuang, Meihua Christina; Kominek, Jacek; Langdon, Quinn K.; Adams, Marie; Koshalek, Justin A.; Hulfachor, Amanda Beth; Opulente, Dana A.; Hall, David J.; Hyma, Katie; Fay, Justin C.; Leducq, Jean-Baptiste; Charron, Guillaume; Landry, Christian R.; Libkind, Diego; Gonçalves, Carla; Gonçalves, Paula; Sampaio, José Paulo; Wang, Qi-Ming; Bai, Feng-Yan; Wrobel, Russel L.; Hittinger, Chris Todd. . Nature Communications. 2023-02-08, 14 (1). doi:10.1038/s41467-023-36139-2.
- He, Peng‐Yu; Shao, Xu‐Qian; Duan, Shou‐Fu; Han, Da‐Yong; Li, Kuan; Shi, Jun‐Yan; Zhang, Ri‐Peng; Han, Pei‐Jie; Wang, Qi‐Ming; Bai, Feng‐Yan. . Yeast. 2022-01, 39 (1-2): 69–82. doi:10.1002/yea.3688.
- Tsai, IJ; Bensasson, D; Burt, A. . Proceedings of the National Academy of Sciences of the United States of America. 2008, 105 (12): 4957–62. Bibcode:2008PNAS..105.4957T. PMC 2290798
 . PMID 18344325. doi:10.1073/pnas.0707314105
. PMID 18344325. doi:10.1073/pnas.0707314105  .
. - Ono, Jasmine; Greig, Duncan. . Current Genetics. 2020-06, 66 (3): 469–474. doi:10.1007/s00294-019-01038-x.
- Pontes, Ana; Čadež, Neža; Gonçalves, Paula; Sampaio, José Paulo. . Frontiers in Genetics. 2019-05-29, 10. doi:10.3389/fgene.2019.00449.
- Boynton, Primrose J.; Greig, Duncan. . Yeast. 2014-10. doi:10.1002/yea.3040.
- Boynton, Primrose J.; Wloch‐Salamon, Dominika; Landermann, Doreen; Stukenbrock, Eva H. . Ecology and Evolution. 2021-06, 11 (11): 6604–6619. doi:10.1002/ece3.7515.
- Leducq, J-B; Charron, G; Samani, P. . Proceedings of the Royal Society B: Biological Sciences. 2014, 281 (1777): 20132472. PMC 3896012
 . PMID 24403328. doi:10.1098/rspb.2013.2472.
. PMID 24403328. doi:10.1098/rspb.2013.2472. - Liti, G; Carter, DM; Moses, AM. . Nature. 2009, 458 (7236): 337–41. Bibcode:2009Natur.458..337L. PMC 2659681
 . PMID 19212322. doi:10.1038/nature07743.
. PMID 19212322. doi:10.1038/nature07743. - Eberlein, Chris; Hénault, Mathieu; Fijarczyk, Anna; Charron, Guillaume; Bouvier, Matteo; Kohn, Linda M.; Anderson, James B.; Landry, Christian R. . Nature Communications. 2019-02-25, 10 (1). doi:10.1038/s41467-019-08809-7.
- Charron, G; Leducq, J-B; Landry, CR. . Molecular Ecology. 2014, 23 (17): 4362–4372. PMID 25039979. S2CID 43071397. doi:10.1111/mec.12864.
- Liti, G; Barton, DB; Louis, EJ. . Genetics. 2006, 174 (2): 839–850. PMC 1602076
 . PMID 16951060. doi:10.1534/genetics.106.062166.
. PMID 16951060. doi:10.1534/genetics.106.062166. - Kuehne, HA; Murphy, HA; Francis, CA. . Current Biology. 2007, 17 (5): 407–11. PMID 17306538. S2CID 18301950. doi:10.1016/j.cub.2006.12.047
 .
. - Zhang, HA; Skelton, A; Gardner, RC. . FEMS Yeast Research. 2010, 10 (7): 941–7. PMID 20868381. doi:10.1111/j.1567-1364.2010.00681.x
 .
. - Naumov, GI; James, SA; Naumova, ES. . International Journal of Systematic and Evolutionary Microbiology. 2000, 50 (5): 1931–42. PMID 11034507. doi:10.1099/00207713-50-5-1931
 .
. - Morales, Lucia; Dujon, Bernard. . Microbiology and Molecular Biology Reviews. 2012-12, 76 (4): 721–739. doi:10.1128/mmbr.00022-12.
- Dunham, Maitreya J; Louis, Edward J. . EMBO reports. 2011-01, 12 (1): 8–10. doi:10.1038/embor.2010.204.
- Dunham, MJ; Louis, ED. . EMBO Reports. 2011, 12 (1): 8–10. PMC 3024138
 . PMID 21151040. doi:10.1038/embor.2010.204.
. PMID 21151040. doi:10.1038/embor.2010.204. - Kellis, Manolis; Patterson, Nick; Endrizzi, Matthew; Birren, Bruce; Lander, Eric S. . Nature. 2003-05, 423 (6937): 241–254. doi:10.1038/nature01644.
- Nikulin, Jarkko; Vidgren, Virve; Krogerus, Kristoffer; Magalhães, Frederico; Valkeemäki, Seija; Kangas-Heiska, Tapio; Gibson, Brian. . European Food Research and Technology. 2020-11, 246 (11): 2283–2297. doi:10.1007/s00217-020-03572-2.
- Costantini, Antonella; Cravero, Maria Carla; Panero, Loretta; Bonello, Federica; Vaudano, Enrico; Pulcini, Laura; Garcia-Moruno, Emilia. . Beverages. 2021-05-28, 7 (2): 30. doi:10.3390/beverages7020030.
- S. Orlić, F.N. Arroyo‐López, K. Huić‐Babić, I. Lucilla, A. Querol, E. Barrio. . Journal of Applied Microbiology. 2010-01, 108 (1): 73–80. doi:10.1111/j.1365-2672.2009.04406.x.
- . Oenobrands.
- Kanter, Jean-Philippe; Benito, Santiago; Brezina, Silvia; Beisert, Beata; Fritsch, Stefanie; Patz, Claus-Dieter; Rauhut, Doris. . Food Chemistry: X. 2020-03, 5: 100072. doi:10.1016/j.fochx.2019.100072.