群体感应
群聚感應()是一種與族群密度有相互關係的刺激和反應的系統。許多細菌會透過群聚感應,根據其族群規模來調節基因的表現。有些社會性昆蟲也會使用和群聚感應的相似方法,決定要在何處建立巢穴。群聚感應除了可以在生態系統當中發揮作用之外,在電腦運算或是機器人的發展上,亦是一項可以應用的技術。
群聚感應可以當作任何分散系統中的決策過程,只要獨立個體有(1)一種方法可評估他們所接觸到的個體的數量和 (2)一旦達到特定閾值的個體所被偵測之後的普遍反應
細菌
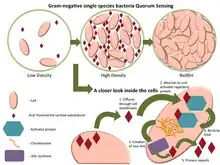
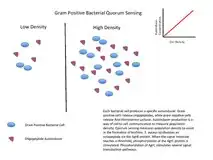
一些群聚感應最有名的例子來自於細菌的研究。細菌利用群體感應協調某些行為,例如根據細菌族群的局部密度的生物膜形成、細菌的毒力,以及細菌對抗生素的抗藥性。群聚感應可以在單一種細菌或是不同的菌種之間發生,且能夠調整多種細菌行為當中的不同步驟。事實上,群聚感應可以簡單的反應細菌族群密度高低,或者是在特定生長環境所對應的擴散速率指標。
許多不同種類的分子可以被用以作為傳遞訊息的信號。最常見的種類包含革蘭氏陽性菌當中的寡肽、存在於革蘭氏陰性菌的N-AHL,以及同時存在於革蘭氏陽性菌與陰性菌、被稱為AI-2的一種自體誘導物[1]。
機制
細菌透過群體感應產生、分泌的傳訊用分子,通常會被稱為自體誘導物或費洛蒙。這些細菌也具有一個可以偵測特定地信號分子(誘導物)的受體。當誘導物接上受體,它可以讓某些基因的轉錄機制被活化,包含用於合成誘導物的那些基因。很少會有細菌能夠偵測到自體分泌的誘導物質。因此,為了活化特定基因的轉錄機制,細胞必須和其他細胞分泌的信號物質相互結合。
當只有少數同類型的其他細菌在附近時,會導致擴散作用減弱,且環繞在附近的誘導物質濃度也會減少(趨近於0)。所以細菌只會製造微量的誘導物。然而,當細菌的族群成長時,誘導物質的濃度會超過閾值,這時會產生更多的誘導物質。如此會形成一個正回饋循環,且受器會被完全的活化。該受器被活化,會誘使其他的特定基因進行「調升」[註 1],而這會幾乎使得所有細胞不約而同地進行轉錄。這種屬於細菌細胞的協調行為在多樣化的情況下是相當有用的。例如:由费氏弧菌(Vibrio fischeri)產生的生物發光,如果是由單一細胞所產生,將是不可見的。當細胞族群很大的時候,藉由群體感應去限制螢光素酶的產生。费氏弧菌的細胞能夠避免浪費能量在生產酶用的產物上。
最近的研究中,對於基因表現活化與停止活化,可以通過實驗進行預測(使細菌族群大小遭受擴散的自體誘導物的訊號的控制)。另一方面,透過這項實驗,能夠發現群體感應下的分子會因為持續的增長,解決一個調節微分方程負擴散係數。
費氏弧菌
群聚感應最早是在费氏弧菌中發現。它是一種寄生於夏威夷短尾魷的生物發光(或是可產生光的器官)互利共生菌[2]。當費雪氏弧菌是自由生活的(像浮游生物的型態),則自體誘發物的濃度會變低,且細胞會因此而不發光。然而,當他們在發光器官中有較高濃度時(大約1011cells/ml),會誘發萤光素酶的轉錄,造成生物發光。
大腸桿菌
在大腸桿菌(E. coli)中,細胞分裂的過程可能部分由AI-2媒介群聚感應調節,lsr 操縱子則為調控此機制的重要因子。部分的 lsr 基因會轉譯為ABC轉運子,在生長曲線初期的延滯階段(latent phase)將AI-2輸入細胞。之後,蛋白激酶LsrK會磷酸化AI-2,磷酸化的AI-2可以內化或用於抑制LsrR。由於LsrR為 lsr 操縱子的抑制劑,故抑制LsrR可激活操縱子。
除了LsrR之外,磷酸二羥丙酮(DHAP)也會與AI-2競爭LsrR的結合位,故能抑制AI-2的作用,達到抑制 lsr 操縱子的效果。甘油醛3-磷酸(G3P)也可通過cAMP-CAPK調節的抑制來抑制操縱子lsr。這解釋了為什麼當葡萄糖變多時,大腸桿菌會失去內化AI-2的能力(因為分解代謝物抑制)。 當正常生長時,AI-2只會暫時存在。
大腸桿菌和沙門氏菌並不製造在其他革蘭氏陰性細菌中常見的AHL信號。然而,他們具有一個可以偵測來自其他細菌AHL信號的受體,在達到一定數量的時候,它們會改變自己的基因表現[3]。
沙門氏菌(Salmonella enterica)
沙門氏菌會表現一種 LuxR 的同源蛋白 SdiA,但並不會轉譯出AHL合成酶。SdiA可偵測由其他細菌種類(包括親水性產氣單胞菌、哈夫尼亞菌和小腸結腸耶氏菌)產生的AHL[4]。當AHL被偵測到,SdiA調節在沙門氏菌致毒性的質體(pefI–srgD– srgA–srgB-rck-srgC)上的操縱子 rck 和一個在染色體 srgE 上的單一基因水平轉移[5][6]。當沙門氏菌通過一些動物的胃腸道時,不會偵測到AHL。這也表明了在正常菌群不會產生AHL。然而,若沙門氏菌在海龜遭受親水性產氣單胞菌寄居,或是大鼠受到小腸結腸炎耶爾森菌感染之時入侵體內,此時即會活化SdiA[7][8]。因此,沙門氏菌似乎使用SdiA去偵測AHL生產其他病原體,而不是偵測正常的腸道菌群。
綠膿桿菌
綠膿桿菌是一種伺機性感染菌,會透過群聚感應機制,調控生物膜的形成、群體運動、外多醣的生產、細菌致毒性以及細胞聚合反應[9]。這些細菌在宿主體內生長且不會傷害到宿主,直到細菌濃度達到閾值後,細菌變成較為強勢,並且發展到可以對抗宿主的免疫系統,並且形成生物膜,導致疾病就像細菌擁有生物膜一樣,有一層保護層在宿主的體內。另一種基因調節的形式,允許細菌通過環境信號快速適應的周圍的變化。最近研究發現厭氧菌相可以顯著影響群體感應的主要調節迴路。群體感應和厭氧生物之間的重要聯繫對該生物體的致毒性的產生具有顯著影響[10]。實驗上,大蒜和人參可以阻斷綠膿桿菌的群體感應[11]。信號分子的治療性酶的降解將阻止這種生物膜的形成並且可能削弱已建立的生物膜。以這種方式中斷信號過程稱為群體感應抑制。
不動桿菌屬
不動桿菌屬(Acinetobacter sp.)最近已被發現,不動桿菌屬也會表現群體感應。這種細菌是一種新的病原菌,會製造AHL[12]。有趣的是不動桿菌屬會同時表現群聚感應和密度感應(quorum quenching)這兩種特性。它會製造AHL也會降解AHL分子[12]。
氣單胞菌
氣單胞菌(Aeromonas sp.)之前被認為是一種魚類的病原菌[13],但是最近發現是人類的病原菌。單孢菌已經能夠在許多病人的感染部位(膽汁、血液、腹腔液、膿液、糞便和尿液)中分離出來。所有被分離出來的菌株皆生產出兩種主要的AHL(C4-HSL和C6-HSL)。有記錄指出溫和氣單胞菌可以製造C6-HSL以及另外兩個具有N-酰基側鍊長於C6的AHL[14]。
耶爾森氏菌
由γ-變形菌綱的小腸結腸炎耶爾森菌(Yersinia enterocolitica)製造的這兩個蛋白質(YenR和YenI)相似於费氏弧菌的LuxR和Lux[15]I[16]。YenR促使非編碼的小核糖核酸(YenS)的表現。YenS抑制YenI的表現和AHL的製造。YenR、YenI、YenS參與了游泳和成群移動的運動能力的控制[15][17]。
與群體感應相關的分子
2001年,首次發表了涉及群聚感應的蛋白質的三維結構;亦於該年,三種LuxS直向同源物的晶體結構藉由X射線也被確定[18]。2002年,哈維氏弧菌的受器LuxP的晶體結構和它的誘導物AI-2(為少數含硼的生物分子)與他結合也被確定了[19]。很多種細菌,包含大腸桿菌及革蘭式陰性細菌的模式生物,都生產AI-2。一個比較基因體學和細菌、古細菌和真核生物的138個基因體樹狀圖演化分析發現酵素LuxS需要AI-2的合成普遍於細菌中。然而,胞質接上結合蛋白LuxP只存在於弧菌菌株導致的結果不是其他生物體可能使用不同於弧菌菌株的AI-2的訊號傳導系統的成分進行感應AI-2的信號,就是他們並沒有如同群聚感應系統[20]。金合歡醇是由真菌白色念珠菌作為一個抑制絲狀的群體感應分子使用[21]。
註解
- 某物質受到特定因素影響引發的後續反應,或產生特定產物。
參考文獻
- Miller, M.B.; Bassler, B.L. . Annu. Rev. Microbiol. 2001, 55 (1): 165–99. PMID 11544353. doi:10.1146/annurev.micro.55.1.165.
- Nealson, K.; Platt, T.; Hastings, J.W. . Journal of Bacteriology. 1970, 104 (1): 313–22 [2018-05-22]. PMC 248216
 . PMID 5473898. (原始内容存档于2009-07-23).
. PMID 5473898. (原始内容存档于2009-07-23). - Ahmer, B.M. . Mol. Microbiol. May 2004, 52 (4): 933–45. PMID 15130116. doi:10.1111/j.1365-2958.2004.04054.x.
- Michael, B.; Smith, J.N.; Swift, S.; Heffron, F.; Ahmer, B.M. . J. Bacteriol. October 2001, 183 (19): 5733–42. PMC 95466
 . PMID 11544237. doi:10.1128/JB.183.19.5733-5742.2001.
. PMID 11544237. doi:10.1128/JB.183.19.5733-5742.2001. - Ahmer, B.M.; van Reeuwijk, J.; Timmers, C.D.; Valentine, P.J.; Heffron, F. . J. Bacteriol. March 1998, 180 (5): 1185–93. PMC 107006
 . PMID 9495757.
. PMID 9495757. - Smith, J.N.; Ahmer, B.M. . J. Bacteriol. February 2003, 185 (4): 1357–66. PMC 142872
 . PMID 12562806. doi:10.1128/JB.185.4.1357-1366.2003.
. PMID 12562806. doi:10.1128/JB.185.4.1357-1366.2003. - Smith, J.N.; Dyszel, J.L.; Soares, J.A.; et al. Ausubel, Frederick M. , 编. . PLoS ONE. 2008, 3 (7): e2826. PMC 2475663
 . PMID 18665275. doi:10.1371/journal.pone.0002826.
. PMID 18665275. doi:10.1371/journal.pone.0002826. - Dyszel, J.L.; Smith, J.N.; Lucas, D.E.; et al. . J. Bacteriol. January 2010, 192 (1): 29–37. PMC 2798265
 . PMID 19820103. doi:10.1128/JB.01139-09.
. PMID 19820103. doi:10.1128/JB.01139-09. - Lewis Sauer, K.; Camper, A.; Ehrlich, G.; Costerton, J.; Davies, D. . Journal of Bacteriology. 2002, 184 (4): 1140–54. ISSN 0021-9193. PMC 134825
 . PMID 11807075. doi:10.1128/jb.184.4.1140-1154.2002.
. PMID 11807075. doi:10.1128/jb.184.4.1140-1154.2002. - Cornelis, P. (ed.). 1st. Caister Academic Press. 2008 [2018-05-22]. ISBN 1-904455-19-0. (原始内容存档于2016-09-12).
- Thomas Bjarnsholt; Peter Østrup Jensen; Thomas B. Rasmussen; Lars Christophersen; Henrik Calum; Morten Hentzer; Hans-Petter Hougen; Jørgen Rygaard; Claus Moser; Leo Eberl; Niels Høiby & Michael Givskov. . Microbiology. 2005, 151 (4): 3873–80. PMID 16339933. doi:10.1099/mic.0.27955-0.
- Kok Gan, Chan; Atkinson, Steve; Mathee, Kalai; Sam, Choon-Kook; Chhabra, Siri Ram; Camara, Miguel; Koh, Chong-Lek & Williams, Paul. . BMC Microbiology. 2011, 11 (1): 51 [2018-05-22]. PMC 3062576
 . PMID 21385437. doi:10.1186/1471-2180-11-51. (原始内容存档于2015-12-05).
. PMID 21385437. doi:10.1186/1471-2180-11-51. (原始内容存档于2015-12-05). - Igbinosa, IH; Igumbor, EU; Aghdasi, F; Tom, M; Okoh, AI. . ScientificWorldJournal. 2012, 2012: 625023. PMC 3373137
 . PMID 22701365. doi:10.1100/2012/625023.
. PMID 22701365. doi:10.1100/2012/625023. - Kok Gan, Chan; Puthucheary, Savithri D.; Chan, Xin-Yue; Yin, Wai-Fong; Wong, Cheng-Siang; Too, Wah-Seng See & Chua, Kek-Heng. . Current Microbiology. 2010, 62 (1): 167–72 [2018-05-22]. PMID 20544198. doi:10.1007/s00284-010-9689-z. (原始内容存档于2019-09-13).
- Atkinson, S; Chang, CY; Sockett, RE; Cámara, M; Williams, P. . Journal of Bacteriology. February 2006, 188 (4): 1451–61. PMC 1367215
 . PMID 16452428. doi:10.1128/JB.188.4.1451-1461.2006.
. PMID 16452428. doi:10.1128/JB.188.4.1451-1461.2006. - Throup, JP; Camara, M; Briggs, GS; Winson, MK; Chhabra, SR; Bycroft, BW; Williams, P; Stewart, GS. . Molecular Microbiology. July 1995, 17 (2): 345–56. PMID 7494483. doi:10.1111/j.1365-2958.1995.mmi_17020345.x.
- Tsai, CS; Winans, SC. . Molecular Microbiology. April 2011, 80 (2): 556–71. PMID 21362062. doi:10.1111/j.1365-2958.2011.07595.x.
- Lewis, H.A.; Furlong, E.B.; Laubert, B.; Eroshkina, G.A.; Batiyenko, Y.; Adams, J.M.; Bergseid, M.G.; Marsh, C.D.; Peat, T.S.; Sanderson, W.E.; Sauder, J.M.; Buchanan, S.G. . Structure. 2001, 9 (6): 527–37. PMID 11435117. doi:10.1016/S0969-2126(01)00613-X.
- Chen, X.; Schauder, S.; Potier, N.; Van Dorsselaer, A.; Pelczer, I.; Bassler, B.; Hughson, F. (PDF). Nature. 2002, 415 (6871): 545–9. PMID 11823863. doi:10.1038/415545a. (原始内容 (PDF)存档于2004年6月22日).
- Sun, J.; Daniel, R.; Wagner-Döbler, I.; Zeng, A.P. . BMC Evol. Biol. 2004, 4 (1): 36 [2018-05-22]. PMC 524169
 . PMID 15456522. doi:10.1186/1471-2148-4-36. (原始内容存档于2015-09-23).
. PMID 15456522. doi:10.1186/1471-2148-4-36. (原始内容存档于2015-09-23). - Jacob M. Hornby. . asm.org. [29 July 2015]. (原始内容存档于2019-02-03).
- Wynendaele, E; Bronselaer, A; Nielandt, J; D'Hondt, M; Stalmans, S; Bracke, N; Verbeke, F; Van De Wiele, C; De Tré, G; De Spiegeleer, B. . Nucleic Acids Research. 2012, 41: 1–5. PMC 3531179
 . PMID 23180797. doi:10.1093/nar/gks1137.
. PMID 23180797. doi:10.1093/nar/gks1137. - . Biopolymers. Sep 2015, 104 (5): 544–51. doi:10.1002/bip.22649.
- O'Loughlin, CT.; et al. . PNAS. 2013, 110 (44): 17981–6. doi:10.1073/pnas.1316981110.
- Norizan, S.; Chan, K. . Sensors. 2013, 13 (4): 5117–5129. doi:10.3390/s130405117.
延伸閱讀
- Dedicated issue of Philosophical Transactions B on quorum sensing. Some articles are freely available.
外部連結
- The Quorum Sensing Website (页面存档备份,存于)
- Cell-to-Cell Communication in Bacteria (页面存档备份,存于)
- The SECOAS project—Development of a Self-Organising, Wireless Sensor Network for Environmental Monitoring
- Measurement of Space: From Ants to Robots
- Instant insight (页面存档备份,存于) into quorum sensing from the Royal Society of Chemistry
- Bonnie Bassler: Discovering bacteria's amazing communication system (页面存档备份,存于)
- Bonnie Bassler's seminar: "Cell-Cell Communication in Bacteria" (页面存档备份,存于)